In u prucessu di fà studii genetichi, spessu scontru insufficiente campioni di RNA, per esempiu, per studià tumuri orali anatomici minusculi, ancu campioni unicellulari, è campioni di mutazioni genetiche specifiche chì sò trascritte à livelli assai bassu in cellule umane.Di sicuru, per a prova COVID-19, se i tamponi ùn sò micca in u locu ghjustu o micca abbastanza volte durante u campionamentu, a dimensione di a mostra serà assai bassa, per quessa chì a Cummissione di Salute è Pianificazione Famiglia hè ghjunta dui ghjorni fà è hà passatu a prova, è se u sampler di l'acidu nucleicu ùn hà micca pigliatu sei campioni, pudete signalà.
A sensibilità di u reagentu hè impurtante perchè avemu stu prublema o quellu prublema, allora chì pudemu fà per migliurà a sensibilità di RT-PCR?
Prima di discutiri e pussibuli suluzioni, mencionemu dui grandi cumplicazioni cù a situazione chì avemu appena citatu.
Prima di tuttu, ci preoccupemu di a perdita di RNA quandu avemu solu uni pochi di pupulazioni di cellule in a nostra mostra.Se i metudi tradiziunali di separazione è di pulizia sò usati, cum'è u metudu di colonna o u metudu di precipitazione di l'acidu nucleicu, ci hè una alta pussibilità chì i pochi mostri seranu persi.Una suluzione hè di aghjunghje una molècula di trasportatore, cum'è tRNA, ma ancu allora, ùn ci hè micca guarantisci chì u nostru esperimentu di ricuperazione hè OK.
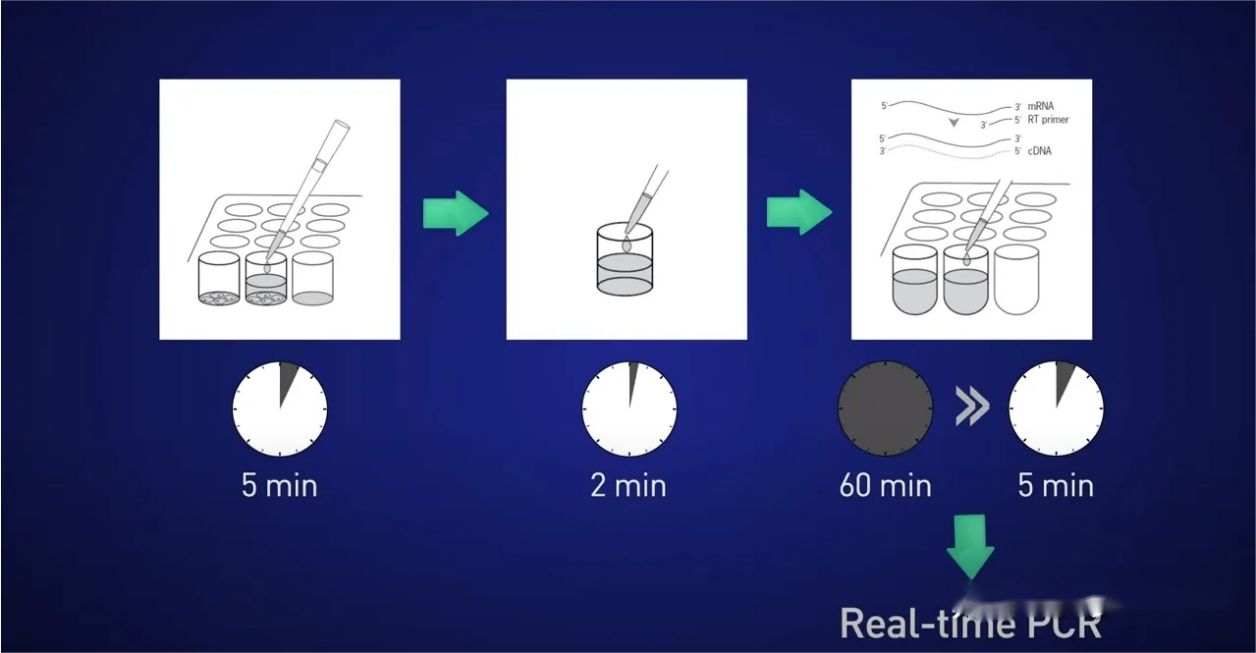
Allora chì hè un modu megliu?Una bona opzione per e cellule cultivate o campioni microanatomici hè di utilizà a lisi diretta.
L'idea hè di split the cells for 5 minutes, liberà l'RNA in a suluzione, poi ferma a reazione per 2 minuti, poi aghjunghje u lisatu direttamente à a reazione di trascrizzione inversa per ùn perde micca RNA, è infine mette u cDNA risultatu direttamente. in a reazione in tempu reale.
Ma chì se, per via di un puntu di partenza limitatu o di una piccula quantità di espressione genica di destinazione, pudemu riciclà tuttu l'RNA è ùn furnisce micca abbastanza mudelli per uttene un bon signalu in tempu reale?
In questu casu, u passu di pre-amplificazione pò esse assai utile.
U seguitu hè un schema per aumentà a sensibilità dopu a trascrizione inversa.Prima di principià, ci vole à dumandà à a valle di quali miri chì ci interessanu, in modu di disignà primers specifichi per questi miri per a pre-amplificazione.
Questu pò esse ottinutu creendu un primer mistu cù finu à 100 pariglii di primers è un ciculu di reazione di 10 à 14 volte.Dunque, un Master Mix cuncepitu specificamente per questu requisitu hè necessariu per pre-amplify l'ADNc ottenutu.
U mutivu di stabilisce u nùmeru di ciculi trà 10 è 14 hè chì stu numaru limitatu di ciculi assicura l'aleatoriu trà e diverse miri, chì hè cruciale per i circadori chì necessitanu infurmazione moleculare quantitativa.
Dopu a pre-amplificazione, pudemu avè una grande quantità di cDNA, cusì chì a sensibilità di deteczione in u back-end hè assai migliurata, è pudemu ancu diluisce a mostra è eseguisce parechje reazzioni PCR in tempu reale per eliminà i pussibuli errori casuali.
Tempu di post: Apr-11-2023